Этот результат указывает нам на ключевую трудность в работе с древней ДНК. Для того чтобы выделить ее из образцов, содержащих очень малое количество сохранного генетического материала, нам нужен очень чувствительный и эффективный метод. Но чем чувствительнее и эффективнее метод, тем с большей вероятностью он дает ложноположительные результаты.
В этих экспериментах ученые использовали полимеразную цепную реакцию (ПЦР) для амплификации (создания копий) ДНК насекомых (рис. 6). ПЦР была разработана в 1983 году Кэри Муллисом, который тогда работал биохимиком в компании Cetus Corporation. Технологии секвенирования ДНК сделали возможным определение точной последовательности нуклеотидов в участке ДНК. Однако эти технологии требовали миллионов клональных копий целевого фрагмента. До изобретения ПЦР этого добивались с помощью бактерий, которые стимулировали захватывать и встраивать в свой геном случайные фрагменты ДНК. Затем из этих бактерий вырастали колонии, в которых каждая бактериальная клетка содержала идентичную копию случайно захваченного фрагмента ДНК: в результате получалось достаточное количество копий, чтобы секвенировать этот участок. ПЦР представляет собой намного более быстрый способ копирования ДНК, а кроме того, и это важнее, она позволяет целенаправленно выбирать участки генома, которые мы будем копировать. Сейчас ПЦР – один из наиболее широко используемых и фундаментальных методов молекулярной биологии.
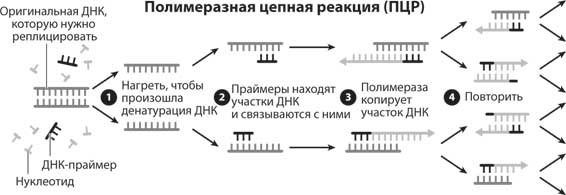
Рис. 6. Полимеразная цепная реакция, или ПЦР. ПЦР – распространенный метод молекулярной биологии, используемый для создания миллиардов копий фрагментов ДНК путем повторного нагревания и охлаждения ДНК в присутствии ДНК-полимеразы (фермента, участвующего в репликации ДНК), при этом из свободных нуклеотидов строятся копии цепочек ДНК и ДНК-праймеры, определяющие, какая часть генома должна быть скопирована
Полимеразная цепная реакция удивительно проста, учитывая революционные последствия ее появления. Чтобы представить себе этот процесс, вообразите, что мы хотим лучше разобраться в генетических различиях между домашними и дикими курами. Считается, что в одомашнивании кур важную роль сыграл ген рецептора тиреотропного гормона (рТТГ), ускоряющий размножение этих птиц. Мы предлагаем использовать ПЦР для амплификации (создания копий) этого гена, извлеченного из ДНК домашней курицы, и из сохранившихся останков древних кур, живших во времена, когда этих птиц еще не одомашнили. Затем мы секвенируем результаты ПЦР, чтобы определить последовательность нуклеотидов в этом гене и выяснить, отличается ли этот ген у домашних кур и их диких родственников и предков, живших в эпоху, предшествовавшую одомашниванию.
Вначале мы должны каким-то образом определить целевой фрагмент ДНК, соответствующий гену рТТГ. Для этого мы создаем ДНК-зонды, называемые праймерами, которые соответствуют участкам ДНК, примыкающим с двух сторон к гену рТТГ. Затем мы создаем смесь, содержащую эти праймеры, предварительно выделенную ДНК курицы, свободные азотистые основания и полимеразу – фермент, работа которого заключается в копировании ДНК. Теперь мы можем начать процесс копирования. Мы нагреваем нашу смесь, чтобы разрушить водородные связи, соединяющие вместе две нити ДНК. Когда молекула полностью разделяется на две нити, мы повторно охлаждаем смесь, вследствие чего они снова соединяются вместе. Поскольку праймеры имеют небольшую длину и в растворе их много, в первую очередь происходит присоединение двух праймеров к тем участкам генома, которым они должны соответствовать, – участкам, примыкающим к гену рТТГ, – и формируется двухцепочечная ДНК на этом участке генома курицы. Наконец, под воздействием полимеразы между праймерами достраивается недостающий участок – ген рТТГ. При этом одна цепочка ДНК выступает в роли шаблона, а свободные нуклеотиды достраиваются к ней, дополняя недостающую последовательность. По окончании этого процесса число копий гена рТТГ удваивается. Чтобы получить достаточное число копий для секвенирования, мы повторяем весь цикл 30–40 раз в течение нескольких часов и в итоге получаем триллионы идентичных копий гена рТТГ.
ПЦР имеет невероятную чувствительность. Теоретически ПЦР сработает, даже если в смеси будет присутствовать только одна копия целевой последовательности ДНК. С одной стороны, это очень хорошо для исследования древней ДНК, где мы должны рассчитывать на очень малое количество сохранившихся участков. С другой стороны, это может привести к катастрофе. Если можно амплифицировать ДНК, имея всего один ее фрагмент, значит, достаточно одного фрагмента ДНК, попавшего из окружающей среды, чтобы сорвать весь эксперимент. Учитывая эту крайне высокую чувствительность к контаминации, уникальные результаты, к примеру обнаружение ДНК насекомых, миллионы лет пробывших в янтаре, потребуют уникального подтверждения своей подлинности. Как минимум, для начала нужно повторить этот результат. В случае эксперимента на курах, который я описала выше, идентичные эксперименты проводились в лабораториях исследования древней ДНК в Даремском университете в Великобритании и Уппсальском университете в Швеции. В этих идентичных экспериментах были получены идентичные результаты секвенирования ДНК из останков древней курицы. Соответственно, результаты первого эксперимента действительно не были следствием контаминации.
Основным источником контаминации при исследовании древней ДНК является ДНК организмов, живущих в настоящее время. ДНК можно обнаружить везде. Она на лабораторной посуде, она в реагентах и растворах, используемых для выделения ДНК. Она на лабораторных столах, полах, стенах и потолках. Она летает по воздуху в лабораториях и коридорах. Что еще хуже, эта современная ДНК находится в прекрасном состоянии с точки зрения физики и химии. В то время как древняя ДНК, как правило, разделена на очень мелкие фрагменты, менее 100 пар оснований на одном целом участке (представьте это в виде слов «кот», «бык», «жук»), нити ДНК современных организмов могут состоять из миллионов пар оснований (представьте это в виде слова «превысокомногорассмотрительствующий»). К тому же древняя ДНК местами разрушена. Фрагментам древней ДНК зачастую недостает азотистых оснований, или же химическая структура этих оснований нарушена («кит», «^ык», «жк»). Фермент полимераза, используемый в ПЦР, не может «прочитать» эти поврежденные участки, и в результате при копировании цепочки появляются ошибки («кут», «зык», «жак»). Еще больше усложняет дело то, что фрагменты древней ДНК зачастую химически связаны с другими участками ДНК, образуя запутанные молекулярные структуры, которые полимераза не опознает как ДНК. Вследствие всех этих проблем полимераза в первую очередь обнаружит чистую, неповрежденную, свободно плавающую, целую контаминантную ДНК и свяжется с ней, предпочтя ее разорванной, соединенной с другими молекулами, поврежденной древней ДНК. Один-единственный фрагмент ДНК живого организма потенциально может вытеснить в ходе ПЦР многие сотни поврежденных фрагментов древней ДНК, что приведет к якобы секвенированию древней ДНК, выделенной из кусочка янтаря. Или из кости мамонта.
Контаминация не пустая угроза. Контаминация может принимать самые разные формы, и она сыграла немалую роль в формировании облика исследований древней ДНК. Первые и единственные участки ДНК динозавров, о которых сообщали исследователи, были (не стоит падать в обморок) результатом контаминации. Многие из них оказались участками человеческой ДНК. Поскольку никто не поверил, что динозавры состояли в более близком родстве с млекопитающими, нежели с птицами или рептилиями, и почти никто не поверил, что ДНК может сохраниться в настолько древних останках динозавров (представлявших собой в большей степени камни, нежели кости), этот результат отклонили, поскольку он определенно был вызван контаминацией.